Ben je geïnteresseerd in de wereld van wetenschap & technologie en wil je hier graag meer over lezen? Word dan lid van KIJK!
Met de nu al honderdduizenden ingevoerde eiwitvormen, zou de nieuwe ‘eiwitalmanak’ onderzoek van over de hele wereld een flinke boost geven.
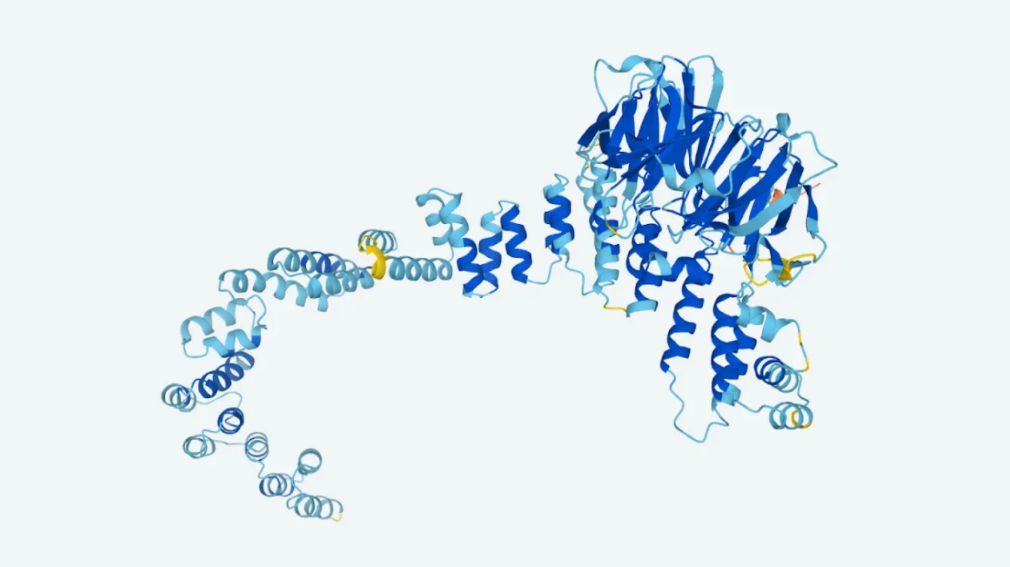
In 2018 maakte het kunstmatige intelligentie-systeem AlphaFold, onderdeel van Google’s DeepMind, zijn debuut bij CASP: een tweejaarlijkse ‘wedstrijd’ waarbij onderzoekers proberen met behulp van software eiwitvormen te voorspellen. Een interessante bijeenkomst, want voorspellen hoe een keten van aminozuren zich zal vouwen tot een eiwit wordt gezien als een soort heilige graal in de biologie.
AlphaFold wist in 2018 in bijna 60 procent van de gevallen de razend complexe eiwitvouwing goed te voorspellen. Vorig jaar werd dat zelfs 90 procent. In twee publicaties in vakblad Nature beschrijven de onderzoekers achter het systeem nu hoe het werkt. Daarbij maakten ze de voorspelde structuren in een database vrij toegankelijk voor de wetenschappelijke wereld: de AlphaFold Protein Structure Database.
Lees ook: A.I. voorspelt 3D-eiwitvouwing
Complexe vouwing
Eiwitten zijn een van de belangrijkste groepen moleculen in ons lichaam. Ze zijn opgebouwd uit lange ketens aminozuren en vervullen afhankelijk van hun vorm een scala aan verschillende taken; van het versnellen van chemische reacties tot het opruimen van ziekteverwekkers.
Hoe zo’n aminozuurketen krult en vouwt tot een eiwit hangt af van de lengte en samenstelling van de reeks. Een minuscuul foutje in de vouwing kan er al voor zorgen dat het eiwit zijn taak minder goed of helemaal niet kan volbrengen. Met alle (negatieve) gevolgen van dien.
Als we op basis van de samenstelling van een eiwit de 3D-structuur kunnen voorspellen, kunnen onderzoekers uitvogelen wat die eiwitten doen, maar ook hoe ze in de knoop raken en zo bijvoorbeeld bepaalde aandoeningen veroorzaken.
Eiwitalmanak
Helaas is dat voorspellen niet makkelijk. Met twintig aminozuren om uit te ‘kiezen’, zijn er gigantisch veel manieren waarop een doorsnee eiwit van een paar honderd bouwsteentjes kan vouwen. Deze kwestie staat al ruim vijftig jaar bekend als het eiwitvouwprobleem.
Gelukkig helpt het kunstmatige intelligentie-systeem AlphaFold wetenschappers bij dit probleem dus uit de brand. Na jaren van ‘oefenen’ is het systeem zo goed geworden in het voorspellen, dat ontwikkelaars DeepMind en EMBL-EBI de zogenaamde AlphaFold Protein Structure Database hebben gelanceerd.
De eerste ‘versie’ van de database bevat pakweg 350.000 eiwitvormen. Het beslaat het menselijk proteoom en die van twintig andere ‘wetenschappelijk relevante organismen’ zoals E. coli, fruitvliegjes en muizen. In de komende maanden hoopt DeepMind de database nog verder uit te breiden. Het doel is uiteindelijk alle meer dan 100 miljoen momenteel bekende eiwitten aan de eiwitalmanak toe te voegen.
Bronnen: Nature1, Nature2, DeepMind
Beeld: AlphaFold/DeepMind